 本文是“漫谈 Clustering 系列”中的第 7 篇,参见本系列的其他文章。
本文是“漫谈 Clustering 系列”中的第 7 篇,参见本系列的其他文章。
由于总是有各种各样的杂事,这个系列的文章竟然一下子拖了好几个月,(实际上其他的日志我也写得比较少),现在决定还是先把这篇降维的日志写完。我甚至都以及忘记了在这个系列中之前有没有讲过“特征”(feature)的概念了,这里不妨再稍微提一下。机器学习应用到各个领域里,会遇到许多不同类型的数据要处理:图像、文本、音频视频以及物理、生物、化学等实验还有其他工业、商业以及军事上得到的各种数据,如果要为每一种类型的数据都设计独立的算法,那显然是非常不现实的事,因此,机器学习算法通常会采用一些标准的数据格式,最常见的一种格式就是每一个数据对应欧几里德空间里的一个向量。
如果原始的数据格式不兼容,那么就需要首先进行转换,这个过程通常叫做“特征提取”(Feature Extraction),而得到的标准数据格式通常叫做 Feature 。例如,一个最简单的将一个文本 Document 转化为向量的方法如下:
- 选定特征空间,这里采用三维欧氏空间,三个维度(依次)分别由 to 、be 和 the 表示。
- 假设待提取的文档是“To be, or not to be: that is the question:”,首先对其进行一些预处理,例如去掉单词的时态后缀、滤掉标点符号等,得到“to be or not to be that be the question”。
- 统计三个维度所对应的单词出现的频率:to 2 次,be 3 次,the 1 次。
- 该文档对应的向量即
[2, 3, 1]。
当然,在实际中我们几乎不会这样人工设定空间的各个维度所对应的单词,而通常是从一个数据集中统计出所有出现的词,再将其中的一些挑选出来作为维度。怎样挑选呢?最简单的办法是根本不做任何挑选,或者简单地只是把出现频率太低的单词(维度)去掉。
不过,事实上我们通常会做更复杂一些的处理,例如,如果你是在做 sentiment analysis ,那么你通常会更加关注语气很重的词,比如 “bad”、“terrible”、“awesome” 等的重要性就比普通的词要大,此时你可以为每一个维度设一个权重,例如,给 “bad” 设置权重 2 ,那么出现 3 次的话,向量在该维度对应的值就为 2*3 = 6 。当然这样手工指定权重只在小范围内可行,如果要为数百万个维度指定权重,那显然是不可能的,另一个稍微自动一点的办法是 tf-idf 。
tf 就是 Term Frequency ,就和刚才说的单词出现的次数差不多,而 idf 则是 Inverse Document Frequency ,通常使用如下公式进行计算:
\[ \displaystyle \text{idf}_t = \log \frac{|D|}{|\{d: t\in d\}|} \]这相当于自动计算出来的每个单词的权重,其想法很简单:如果在许多文档中都出现的词(例如虚词、语气词等),它所包含的信息量通常会比较小,所以用以上的公式计算出来的权重也会相对较小;另一方面,如果单词并不是在很多文档中都出现的话,很有可能就是出现的那些文档的重要特征,因此权重会相对大一些。
前面说了一堆,其实主要是想要对 feature 做一些“预”处理,让它更“好”一些,手工设置维度的权重固然是很人力,其实 tf-idf 也是在假定了原始 feature 是 document 、term 这样的形式(或者类似的模型)的情况下才适用的(例如,在门禁之类的系统里,feature 可能有声音、人脸图像以及指纹等数据,就不能这样来处理),因此也并不能说是一种通用的方法。
然而,既然机器学习的算法可以在不考虑原始数据的来源和意义的情况下工作,那么 feature 的处理应该也可以。事实也是如此,包括 feature selection 和 dimensionality reduction 两个 topic 都有许多很经典的算法。前者通常是选出重要的 feature 的维度(并抛弃不重要的维度),而后者则是更广泛意义上的把一个高维的向量映射为一个低维向量,亦即“降维”,得到的结果 feature 的值已经不一定是原始的值,也可以把 feature selection 看作是 dimensionality reduction 的一种特殊情况。举一个例子,tf-idf 实际上不算 feature selection ,因为它(通常)并没有丢弃低权值的维度,并且处理过后的特征的每个维度都被乘上了一个权值,不再是原来的值了;但是它却可以被看作一种降维,虽然严格意义上来说维度并没有“降低”。简单来说降维可以看作一个函数,其输入是一个 D 维的向量,输出是一个 M 维的向量。
按照机器学习的方法的一贯作风,我们需要定义目标函数并进行最优化。不同的目标也就导致了不同的降维算法,这也正是今天要讲的话题。
然而,我们的目的究竟是什么呢?一个比较直接的问题是原始的数据量维度太高,我们无法处理,因此需要降维,此时我们通常希望在最大限度地降低数据的维度的前提下能够同时保证保留目标的重要的信息,就好比在做有损的数据压缩时希望压缩比尽量大但是质量损失不要太多。于是问题又转化为如何衡量对信息的保留程度了。
一个最直接的办法就是衡量 reconstruction error ,即
\[ \displaystyle \frac{1}{N}\sum_{i=1}^N \|\mathbf{x}_i-\tilde{\mathbf{x}}_i\|^2 \]其中 $\tilde{\mathbf{x}}_i$ 是 $\mathbf{x}_i$ 所对应的低维表示再重新构造出来的高维形式,就相当于是压缩之后解压出来的结果,不过虽然有许多压缩方法都是无损的,就是说这个差值会等于零,但是大部分降维的结果都是有损的。不过我们仍然希望把上面的 reconstruction error 最小化。
另外一种方式是简单地使用 variance 来衡量所包含信息量,例如,我们要把一些 D 维的向量降为 1 维,那么我们希望这一维的 variance 达到最大化,亦即:
\[ \displaystyle \arg \max_f \frac{1}{N}\sum_{i=1}^N \left(f(\mathbf{x}_i)-\overline{f(\mathbf{x}_i)}\right)^2 \]其中 $f$ 是降维函数。推而广之,如果是降为 2 维,那么我希望第 2 维去关注第 1 维之外的 variance ,所以要求它在与第一维垂直的情况下也达到 variance 最大化。以此类推。
然而,当我们把降维函数 $f$ 限定维线性的时候,两种途径会得到同样的结果,就是被广泛使用的 Principal Components Analysis(PCA) 。PCA 的降维函数是线性的,可以用一个 $D\times M$ 维的矩阵 $W$ 来表示,因此,一个 D 维的向量 $\mathbf{x}$ 经过线性变换 $W^T\mathbf{x}$ 之后得到一个 M 维向量,就是降维的结果。把原始数据按行排列为一个 $N\times D$ 维的矩阵 $X$ ,则 $XW$ 就是降维后的 $N\times M$ 维的数据矩阵,目标是使其 covariance 矩阵最大。在数据被规则化(即减去其平均值)过的情况下,协方差矩阵 (covariance) $S = \frac{1}{N}W^TX^TXW$ ,当然矩阵不是一个数,不能直接最大化,如果我们采用矩阵的 Trace (亦即其对角线上元素的和)来衡量其大小的话,要对 $S$ 求最大化,只需要求出 $X^TX$ 的特征值和特征向量,将 M 个最大的特征值所对应的特征向量按列排列起来组成线性变换矩阵 $W$ 即可。这也就是 PCA 的求解过程,得到的降维矩阵 $W$ 可以直接用到新的数据上。如果熟悉 Latent Semantic Analysis (LSA) 的话,大概已经看出 PCA 和 Singular Value Decomposition (SVD) 以及 LSA 之间的关系了。以下这张图(引自《The Elements of Statistical Learning》)可以直观地看出来 PCA 做了什么,对于一些原始为二维的数据,PCA 首先找到了 variance 最大的那一个方向:
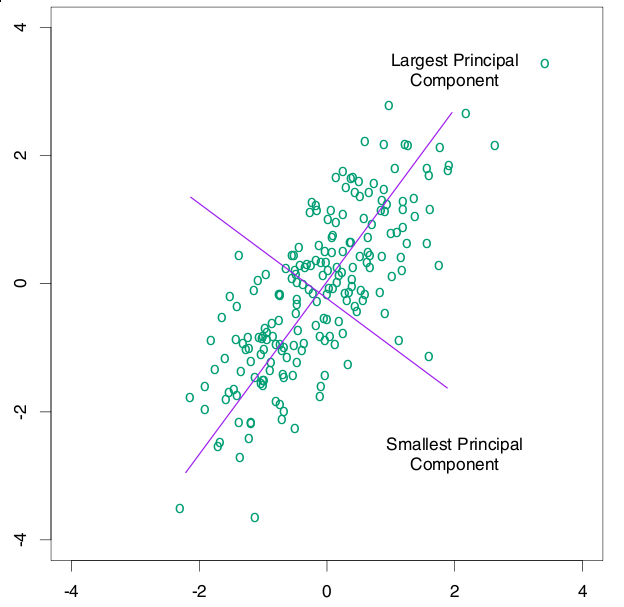
PCA 作为一种经典的降维方法,被广泛地应用于机器学习、计算机视觉以及信息检索等各个领域,其地位类似于聚类中的 K-means ,在现在关于降维等算法的研究中也经常被作为 baseline 出现。然而,PCA 也有一些比较明显的缺点:一个就是 PCA 降维是线性变换,虽然线性变换计算方便,并且可以很容易地推广到新的数据上,然而有些时候线性变换也许并不合适,为此有许多扩展被提出来,其中一个就是 Kernel PCA ,用 Kernel Trick 来将 PCA 推广到非线性的情况。另外,PCA 实际上可以看作是一个具有 Gaussian 先验和条件概率分布的 latent variable 模型,它假定数据的 mean 和 variance 是重要的特征,并依靠 covariance 最大化来作为优化目标,而事实上这有时候对于解决问题帮助并不大。
一个典型的问题就是做聚类或分类,回想我们之前谈过的 Spectral Clustering ,就是使用 Laplacian eigenmap 降维之后再做 K-means 聚类,如果问题定下来了要对数据进行区分的话,“目的”就变得明朗了一些,也就是为了能够区分不同类别的数据,再考虑直观的情况,我们希望如果通过降维把高维数据变换到一个二维平面上的话,可以很容易“看”出来不同类别的数据被映射到了不同的地方。虽然 PCA 极力降低 reconstruction error ,试图得到可以代表原始数据的 components ,但是却无法保证这些 components 是有助于区分不同类别的。如果我们有训练数据的类别标签,则可以用 Fisher Linear Discriminant Analysis 来处理这个问题。
同 PCA 一样,Fisher Linear Discriminant Analysis 也是一个线性映射模型,只不过它的目标函数并不是 Variance 最大化,而是有针对性地使投影之后属于同一个类别的数据之间的 variance 最小化,并且同时属于不同类别的数据之间的 variance 最大化。具体的形式和推导可以参见《Pattern Classification》这本书的第三章 Component Analysis and Discriminants 。
当然,很多时候(比如做聚类)我们并不知道原始数据是属于哪个类别的,此时 Linear Discriminant Analysis 就没有办法了。不过,如果我们假设原始的数据形式就是可区分的的话,则可以通过保持这种可区分度的方式来做降维,MDS 是 PCA 之外的另一种经典的降维方法,它降维的限制就是要保持数据之间的相对距离。实际上 MDS 甚至不要求原始数据是处在一个何种空间中的,只要给出他们之间的相对“距离”,它就可以将其映射到一个低维欧氏空间中,通常是三维或者二维,用于做 visualization 。
不过我在这里并不打算细讲 MDS ,而是想说一下在 Spectral Clustering 中用到的降维方法 Laplacian Eigenmap 。同 MDS 类似,LE 也只需要有原始数据的相似度矩阵,不过这里通常要求这个相似度矩阵 $S$ 具有局部性质,亦即只考虑局部领域内的相似性,如果点 $i$ 和 $j$ 距离太远的话,$S_{ij}$ 应该为零。有两种很直接的办法可以让普通的相似度矩阵具有这种局部性质:
- 通过设置一个阈值,相似度在阈值以下的都直接置为零,这相当于在一个 $\epsilon$-领域内考虑局部性。
- 对每个点选取 k 个最接近的点作为邻居,与其他的点的相似性则置为零。这里需要注意的是 LE 要求相似度矩阵具有对称性,因此,我们通常会在 $i$ 属于 $j$ 的 k 个最接近的邻居且/或反之的时候,就保留 $S_{ij}$ 的值,否则置为零。
构造好 $S$ 之后再来考虑降维,从最简单的情况开始,即降到一维 $\mathbf{x}_i \rightarrow y_i$ ,通过最小化如下目标函数来实现:
\[ \displaystyle \sum_{ij}(y_i-y_j)^2 W_{ij} \]从直观上来说,这样的目标函数的意义在于:如果原来 $\mathbf{x}_i$ 和 $\mathbf{x}_j$ 比较接近,那么 $W_{ij}$ 会相对比较大,这样如果映射过后 $y_i$ 和 $y_j$ 相差比较大的话,就会被权重 $W_{ij}$ 放大,因此最小化目标函数就保证了原来相近的点在映射过后也不会彼此相差太远。
令 $D$ 为将 $W$ 的每一行加起来所得到的对角阵,而 $L = D-W$ ,被称作是拉普拉斯矩阵,通过求解如下的特征值问题
\[ \displaystyle L\mathbf{f} = \lambda D \mathbf{f} \]易知最小的那个特征值肯定是 0 ,除此之外的最小的特征值所对应的特征向量就是映射后的结果。特征向量是一个 N 维列向量,将其旋转一下,正好是 N 个原始数据降到一维之后的结果。
类似地推广到 M 维的情况,只需要取除去 0 之外的最小的 M 个特征值所对应的特征向量,转置之后按行排列起来,就是降维后的结果。用 Matlab 代码写出来如下所示(使用了 knn 来构造相似度矩阵,并且没有用 heat kernel ):
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 | % Laplacian Eigenmap ALGORITHM (using K nearest neighbors) % % [Y] = le(X,K,dmax) % % X = data as D x N matrix (D = dimensionality, N = #points) % K = number of neighbors % dmax = max embedding dimensionality % Y = embedding as dmax x N matrix %%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%% function [Y] = leigs(X,K,d) [D,N] = size(X); fprintf(1,'LE running on %d points in %d dimensions\n',N,D); % STEP1: COMPUTE PAIRWISE DISTANCES & FIND NEIGHBORS fprintf(1,'-->Finding %d nearest neighbours.\n',K); X2 = sum(X.^2,1); distance = repmat(X2,N,1)+repmat(X2',1,N)-2*X'*X; [sorted,index] = sort(distance); neighborhood = index(2:(1+K),:); % STEP2: Construct similarity matrix W fprintf(1,'-->Constructing similarity matrix.\n'); W = zeros(N, N); for ii=1:N W(ii, neighborhood(:, ii)) = 1; W(neighborhood(:, ii), ii) = 1; end % STEP 3: COMPUTE EMBEDDING FROM EIGENVECTS OF L fprintf(1,'-->Computing embedding.\n'); D = diag(sum(W)); L = D-W; % CALCULATION OF EMBEDDING options.disp = 0; options.isreal = 1; options.issym = 1; [Y,eigenvals] = eigs(L,d+1,0,options); Y = Y(:,2:d+1)'*sqrt(N); % bottom evect is [1,1,1,1...] with eval 0 fprintf(1,'Done.\n'); %%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%% |
事实上,Laplacian Eigenmap 假设数据分布在一个嵌套在高维空间中的低维流形上, Laplacian Matrix $L$ 则是流形的 Laplace Beltrami operator 的一个离散近似。关于流形以及 Laplacian Eigenmap 这个模型的理论知识就不在这里做更多地展开了,下面看一个比较直观的例子 Swiss Roll 。
Swiss Roll 是一个像面包圈一样的结构,可以看作一个嵌套在三维空间中的二维流形,如下图所示,左边是 Swiss Roll ,右边是从 Swiss Roll 中随机选出来的一些点,为了标明点在流形上的位置,给它们都标上了颜色。
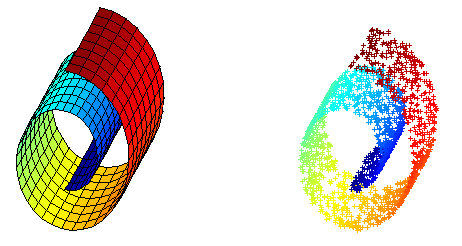
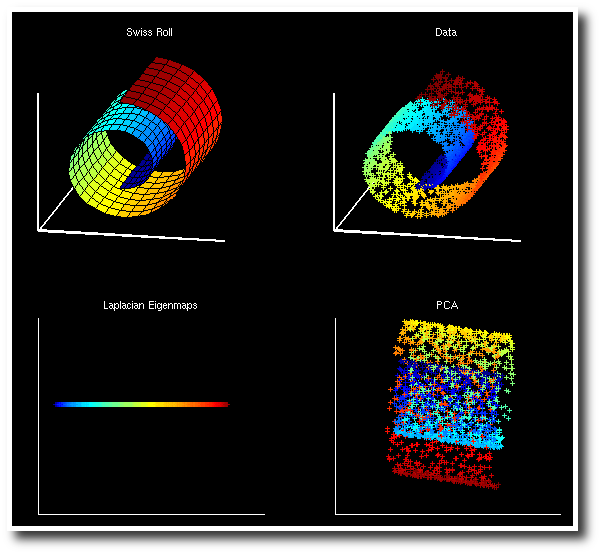
而下图是 Laplacian Eigenmap 和 PCA 分别对 Swiss Roll 降维的结果,可以看到 LE 成功地把这个流形展开把在流形上属于不同位置的点映射到了不同的地方,而 PCA 的结果则很糟糕,几种颜色都混杂在了一起。
另外,还有一种叫做 Locally Linear Embedding 的降维方法,它同 LE 一样采用了流形假设,并假定平滑流形在局部具有线性性质,一个点可以通过其局部邻域内的点重构出来。首先它会将下式最小化
\[ \displaystyle \sum_i \|\mathbf{x}_i - \sum_j W_{ij}\mathbf{x}_j\|^2 \]以求解出最优的局部线性重构矩阵 $W$ ,对于距离较远的点 $i$ 和 $j$ ,$W_{ij}$ 应当等于零。这之后再把 $W$ 当作已知量对下式进行最小化:
\[ \displaystyle \sum_i \|\mathbf{y}_i - \sum_j W_{ij}\mathbf{y}_j\|^2 \]这里 $\mathbf{y}$ 成了变量,亦即降维后的向量,对这个式子求最小化的意义很明显了,就是要求如果原来的数据 $\mathbf{x}_i$ 可以以 $W$ 矩阵里对应的系数根据其邻域内的点重构出来的话,那么降维过后的数据也应该保持这个性质。
经过一些变换之后得到的求解方法和 LE 类似,也是要求解一个特征值问题,实际上,从理论上也可以得出二者之间的联系(LE 对应于 $\mathcal{L}$ 而 LLE 对应于 $\mathcal{L}^2$),如果感兴趣的话,可以参考 Laplacian Eigenmaps for Dimensionality Reduction and Data Representation 这篇论文里的对比。下面是两种方法在 Swiss Roll 数据上的结果,也是非常相似的:
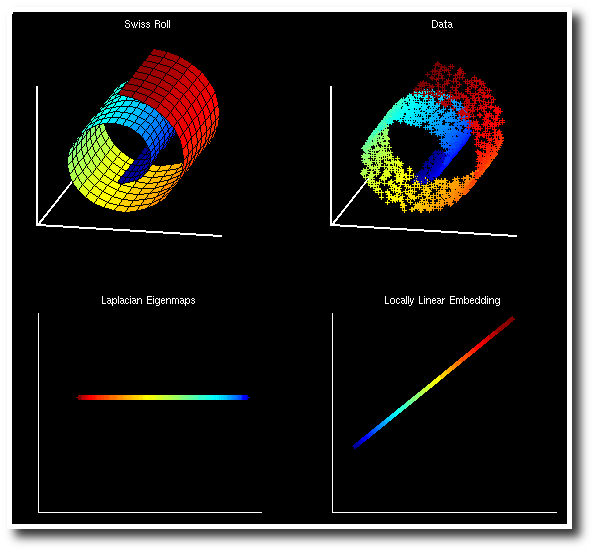
有一点需要注意的是,LE 和 LLE 都是非线性的方法,PCA 得到的结果是一个投影矩阵,这个结果可以保存下来,在之后对任意向量进行投影,而 LE 和 LLE都是直接得出了数据降维之后的结果,但是对于新的数据,却没有得到一个“降维函数”,没有一个合适的方法得到新的数据的降维结果。所以,在人们努力寻求非线性形式扩展 PCA 的时候,另一些人则提出了 LE 和 LLE 的线性形式,分别叫做 Locality Preserving Projection 和 Neighborhood Preserving Embedding 。
在 LPP 中,降维函数跟 PCA 中一样被定为是一个线性变换,用一个降维矩阵 $A$ 来表示,于是 LE 的目标函数变为
\[ \displaystyle \sum_{ij}(A^T\mathbf{x}_i-A^T\mathbf{x}_j)^2 W_{ij} \]经过类似的推导,最终要求解的特征值问题如下:
\[ \displaystyle X^TLX\mathbf{a} = \lambda X^TDX \mathbf{a} \]得到的按照特征值从小到大排序的特征向量就组成映射矩阵 $A$ ,和 LE 不同的是这里不需要去掉第一个特征向量。另一点是在 LE 中的特征值是一个稀疏的特征值问题,在只需要求解最小的几个特征值的时候可以比较高效地求解,而这里的矩阵在乘以 $X$ 之后通常就不再稀疏了,计算量会变得比较大,这个问题可以使用 Spectral Regression 的方法来解决,参见 Spectral Regression: A Unified Approach for Sparse Subspace Learning 这篇 paper 。如果采用 Kernel Trick 再把 LPP 非线性化的话,又会回到 LE 。而 LLE 的线性版本 NPE 也是采用了类似的办法来得到的,就不在这里多讲了。
另外,虽然 LE 是 unsupervised 的,但是如果训练数据确实有标签可用,也是可以加以利用的——在构造相似度矩阵的时候,属于同一类别的相似度要大一些,而不同类别的相似度则会小一些。
当然,除去聚类或分类之外,降维本身也是一种比较通用的数据分析的方法,不过有许多人批评降维,说得到的结果没有意义,不用说非线性,就是最简单的线性降维,除去一些非藏极端的特殊情况的话,通常将原来的分量线性组合一下都不会得到什么有现成的物理意义的量了。然而话也说回来,现在的机器学习几乎都是更 prefer “黑盒子”式的方法吧,比如决策树,各个分支对应与变量的话,它的决策过程其实人是可以“看到”或者说“理解”的,但是 SVM 就不那么“直观“了,如果再加上降维处理,就更加“不透明”了。不过我觉得这没什么不好的,如果只是靠可以清晰描诉出来的 rule 的话,似乎感觉神秘感不够,没法发展出“智能”来啊 ^_^bb 最后,所谓有没有物理意义,其实物理量不过也都是人为了描述问题方便而定义出来的吧。

kid,你不做professor真的可惜了。。。
@zhouyuan
-,-//
深入浅出,问题描述的很清楚
很适合当老师
其实,现在很多降维算法的最终目的已经不是减少维度,从而减少计算量了。而是为了解决Semantic gap的问题。就是说,最终降到几维,可能是不确定的。比如原始数据有300维,我算出来400维的空间中,数据分离的最好,那就到400维好了。所以降维学出那个manifold,基本上任务就结束了……
刚学习降维方面的内容!我现在就认为不同的降维方法就两个地方不同,一是定义对象之间的形似度和相异度的方法不同,另一个就是目标函数不一样。还有关于是否是线性的理解还不是很充分,我总认为线性的就是一个像加权一样,比如一个对象有n个特征,要把它降维为m维,则就是让降维后的每一个特征都是前面n个特征的加权值,不知道我理解对不?能否对我讲下这个线性和非线性本质的区别是什么,我的理解不是很合理!
@heshizhu
差不多吧,线性就是你能得到一个 W ,去乘以原来的 x 就能得到低维的形式,就是加权和了,而非线性的话,就没有一个固定形式或者可以用这样的线性解析式写出来的降维方法了。
“同 PCA 一样,Fisher Linear Discriminant Analysis 也是一个线性映射模型,只不过它的目标函数并不是 Variance 最大化,而是有针对性地使投影之后属于同一个类别的数据之间的 variance 最小化,并且同时属于不同类别的数据之间的 variance 最大化。”
土问:不同类别之间的“variance”是怎么定义来着??
我觉得应该是所有类别的样本总体有一个Variance,(设V_Totle)。各个类别的样本有各自的Variance,(设V_individual)。而类别之间的“variance”是指V_Totle – sigma(V_individual)。这样理解起来比较顺。
options.disp = 0; options.isreal = 1; options.issym = 1;
在matlab里面加这些选项是为了加速特征根的求解么??
土问怎么样按Swiss Roll的分布来随机采样点..大侠轻点拍~~
@Chrysalis
其实 V_{total}-V_{individual} = V_{between} 了,所以用哪种方法来理解都可以,要看个人喜好了…… -.-。
加上一些限制是有可能会加速求解的,因为不同的情况可能会有不同的算法适合,不过具体是怎么影响的我也不了解。
Swiss Roll 那个曲面上随机选点就可以了啊,你搜索一下 matlab swiss roll 会找到很多现成的代码。
@cerror
kid这个是在说什么?? 从300到400维..
[…] 漫谈 Clustering (番外篇): Dimensionality Reduction by pluskid, on 2009-06-20, in Machine Learning 11 comments […]
这行code是不是有问题?
[Y,eigenvals] = eigs(L,d+1,0,options);
等于现在求的是L的eigenvector。
而本来应该求的是 inv(D) .* L 的eigenvector ([latex] Lf = \lambda Df \Rightarrow D^{-1}Lf = \lambda f [/latex])
恩,有好几种变种,最“正确”的做法应该是直接用 eigs 来解 generalized eigenvalue problem ,也就是求 $Lf = \lambda Df$ ,但是不显式地求 D 的逆。我好想也有见过直接求 $L$ 的特征值的,不过如果 Weight 是 normalize 到 1 的话,两种就等价了。
还是不太理解怎么把Weight给normalize成1?之后为什么就只用分解L就行了。
我自己试了inv(D) .* L (D是对角阵,求逆也不慢)
然后手工把eigenvector调整为符合 f^T D f = 1 并且 f^T D 1 = 0 (这个等于0的式子在paper中出现了,没在这里的文章中出现),然后得到的结果并不是一条直线 (http://zhanxw.com/blog/wp-content/uploads/2011/02/LaplacianEigenmap.jpg),不知pluskid碰没碰到过?谢谢。
就是 weight 每一行加起来等于 1 的话,D 就等于 identity 矩阵了。
求逆倒不一定是慢,只是容易出现数值误差。你这个结果是 swiss roll ?不是挺正常的吗?为什么一定要是一条直线?
你说的对:只要把对角阵D的每一个元素设置为1就成单位阵了(我错误的认为D_i = \sum_j W_{ij})。
至于Swiss Roll,我是看了你这篇Blog的最后两个黑底色的图,上面的Laplacian Eigenmap都是一条直线。我想不出来怎么能得到这种结果 🙁
恩,Laplacian Eigenmap 似乎是大部分时候跑出来都是这种弯曲的结果。直条比较难跑,试试各种参数和变种吧,我都忘记自己当时是用什么参数跑出来的了。不过如果仅仅是说结果的话,我觉得这个弯曲的和直条的倒是并不能就说谁好谁坏的啦。
原来你两年前就知道这个了!我最近在用isomap降维MFCCs,因为要计算没每点之间的距离,数据量太大内存不够,3000×13的数据就已经极限了。你知道其他的非线性降维方法的实现会不会不占那么多内存而且没有太慢?
非线性降维一般都是 kernel PCA 一系列的,要算 similarity matrix ,一般点数多了就受不了。像 Laplacian Eigenmaps 之类的,用稀疏矩阵的话,数量级有可以上去一些。而且许多做大规模 spectral clustering 的,应该也就实现了大规模的非线性降维,可以参考一下。
[…] 漫谈 Clustering (番外篇): Dimensionality Reduction […]
看了这篇文章,才知道降维有这么多学问。我以为我在做分类聚类时的特征选取就是降维了呢。不过,我做的都是文本的。
通俗易懂,我收获很大,我有个小问题,麻烦大侠赐教:(NJW算法)原始数据->相似度矩阵->对角矩阵->拉普拉斯矩阵->行归一化->k-means聚类->聚类的索引
我的原始数据是一张灰度图的灰度值矩阵,我的问题是按上面步骤走下来得到聚类的索引后怎样显示聚类后的图像,matlab 里有相应的函数还是有其他方法?毕竟行归一化后的数据早已不是原始数据了。(看到博主连2011年的帖子也回,心里很期望,我大四做毕设,在自学谱聚类,希望博主能帮帮忙,谢谢)
你好,谱聚类能得到的只是聚类标签而已,不知道你说的“聚类后的图像”是什么意思,你是说每一个类的中心对应的那个图像?如果那样的话最简单的方法就是把每一类里所有点求一个平均值,不过如果数据不是类似于球形的簇状分布的话,这样求出来的并不是真正的中心,但如果本身是不规则的形状的话,连“中心”这个概念的定义也都比较麻烦了。
不好意思补充下:行归一化->特征向量矩阵->k-means聚类->聚类的索引
对呀,可以得到每个类的标签和中心,若对一幅图像直接k-means聚类的话,得到每个标签就是该类的所有像素原始数据的索引,但问题是谱聚类中k-means方法针对的不是原始像素数据,而是特征向量组成的矩阵,得到的标签所指示的数据集合应该不是原始数据(像素值)了吧,我的问题是:如果不是原始数据,那怎么得到原始数据,用来作为像素值显示呢?(将原始图像划分为几块,分别显示)matlab有更高级的函数,还是继续写代码将这些标签对应的非原始数据转变为原始数据?
谱聚类得到的结果就是对原始数据点的聚类,如果得到的结果跟输入数据都不对应,那这个算法根本就没有用了啊……
嗯,也是啊,我再继续学学,多谢了
我用作者的LLE代码给Swiss Roll降维的时候,把降后的维度d设为2,也就是降到二维。降维后生成的LLE图跟你文章里面一样是一条直线,也就是一维的。我看了一下降维后的Y矩阵数据,发现有一维所有的数据都是-1,觉得很奇怪。
明明设定降到二维,怎么结果却降到一维去了?
多调一下构建 graph 时候的最近邻个数 k ,有的时候可以展开……
我改了好多k值结果还是直线,后来发现LLE的作者在网站上说因为Matlab的eigs函数在不同的版本不一样,所以用的是matlab5.3以后的版本lle.m里面的
Y = Y(:,2:d+1)’*sqrt(N);
应该改成
Y = Y(:,1:d)’*sqrt(N);
然后结果就是二维的了~
-_-#….
呃……这么神奇的,莫非是新版本的 matlab eigs 会自己把零特征值去掉?看来以后得注意一下这个问题……
零特征值去不去掉有什么影响呢?
零特征值对应的特征向量是全常数向量,相当于那个维度就没有了。
不是很懂。。。我再看看相关知识好了~~ 谢谢Kid :)
和原向量方向一样,不晓得这样是否可以解释
还有请谈谈最大似然估计,谢谢!
博主你好:
请问求矩阵L时
D = diag(sum(W));
L = D-W;
和
D = sum(W, 2) + (1e-10);
D = sqrt(1./D); % D^(-1/2)
D = spdiags(D, 0, n, n);
L = D *W * D;
是一回事不?第二段代码是您在“漫谈clustering(4)”最后推荐的spectral clustering包里sc.m中的相关代码”这两部分代码是各自适应不同情况的similarity matrix吗?
你好,这样得到的是一个不同的 Laplacian 矩阵,Laplacian 矩阵有好几个变种,具体可参加有一篇 Spectral Clustering 的 Tutorial 。
恩,谢了。正在看那个tutorial,突然想起来你前面提到的那句话了:“Usually, every author just calls “his” matrix the graph Laplacian”
我想问的问题很简单,就是LLE算法后得到的结果 可以说是像一条直线一样的,我怎么能得到 一片散点一样的图呢?相应的程序代码是什么呢?
楼主LLE并没仿真出正确结果啊,因为作者网站的matlab代码在低版本的matlab下运行的。有个函数的输出顺序和高版本输出顺序相反,所以运行结果就是楼主这样。
嗯,不同版本 matlab 的 eigs 得到的特征向量的排序变化了。
网址是 http://www.cs.nyu.edu/~roweis/lle/
我想问一下,特征值和特征向量有什么几何意义?还有您有没有研究过稀疏表达和低秩表达呢,非常期待看到楼主关于这两个主题的介绍^_^
东东居然在这里看到你了。。。。
不知道你说的几何意义是什么啊。特征向量可以看成是映射后的低维坐标。 low rank 和 sparse dictionary learning 这些都没有专门研究过呢。
博主,你好!“类似地推广到 M 维的情况,只需要取除去 0 之外的最小的 M 个特征值所对应的特征向量,转置之后按行排列起来,就是降维后的结果。”这样的话,那谱聚类中取的前k个最小特征值,也是除去0之后的前k个吗?
原来你导师是he啊,怪不得,呵呵
请问,有没有关于LLE推导过程的资料呢?