 本文是“漫谈 Clustering 系列”中的第 8 篇,参见本系列的其他文章。
本文是“漫谈 Clustering 系列”中的第 8 篇,参见本系列的其他文章。
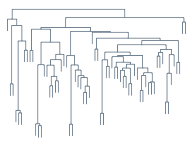
系列不小心又拖了好久,其实正儿八经的 blog 也好久没有写了,因为比较忙嘛,不过觉得 Hierarchical Clustering 这个话题我能说的东西应该不多,所以还是先写了吧(我准备这次一个公式都不贴 😀 )。Hierarchical Clustering 正如它字面上的意思那样,是层次化的聚类,得出来的结构是一棵树,如右图所示。在前面我们介绍过不少聚类方法,但是都是“平坦”型的聚类,然而他们还有一个更大的共同点,或者说是弱点,就是难以确定类别数。实际上,(在某次不太正式的电话面试里)我曾被问及过这个问题,就是聚类的时候如何确定类别数。
我能想到的方法都是比较 naive 或者比较不靠谱的,比如:
- 根据数据的来源使用领域相关的以及一些先验的知识来进行估计——说了等于没有说啊……
- 降维到二维平面上,然后如果数据形状比较好的话,也许可以直观地看出类别的大致数目。
- 通过谱分析,找相邻特征值 gap 较大的地方——这个方法我只了解个大概,而且我觉得“较大”这样的词也让它变得不能自动化了。
当时对方问“你还有没有什么问题”的时候我竟然忘记了问他这个问题到底有没有什么更好的解决办法,事后真是相当后悔啊。不过后来在实验室里询问了一下,得到一些线索,总的来说复杂度是比较高的,待我下次有机会再细说(先自己研究研究)。
不过言归正传,这里要说的 Hierarchical Clustering 从某种意义上来说也算是解决了这个问题,因为在做 Clustering 的时候并不需要知道类别数,而得到的结果是一棵树,事后可以在任意的地方横切一刀,得到指定数目的 cluster ,按需取即可。
听上去很诱人,不过其实 Hierarchical Clustering 的想法很简单,主要分为两大类:agglomerative(自底向上)和 divisive(自顶向下)。首先说前者,自底向上,一开始,每个数据点各自为一个类别,然后每一次迭代选取距离最近的两个类别,把他们合并,直到最后只剩下一个类别为止,至此一棵树构造完成。
看起来很简单吧? 😀 其实确实也是比较简单的,不过还是有两个问题需要先说清除才行:
- 如何计算两个点的距离?这个通常是 problem dependent 的,一般情况下可以直接用一些比较通用的距离就可以了,比如欧氏距离等。
- 如何计算两个类别之间的距离?一开始所有的类别都是一个点,计算距离只是计算两个点之间的距离,但是经过后续合并之后,一个类别里就不止一个点了,那距离又要怎样算呢?到这里又有三个变种:
- Single Linkage:又叫做 nearest-neighbor ,就是取两个集合中距离最近的两个点的距离作为这两个集合的距离,容易造成一种叫做 Chaining 的效果,两个 cluster 明明从“大局”上离得比较远,但是由于其中个别的点距离比较近就被合并了,并且这样合并之后 Chaining 效应会进一步扩大,最后会得到比较松散的 cluster 。
- Complete Linkage:这个则完全是 Single Linkage 的反面极端,取两个集合中距离最远的两个点的距离作为两个集合的距离。其效果也是刚好相反的,限制非常大,两个 cluster 即使已经很接近了,但是只要有不配合的点存在,就顽固到底,老死不相合并,也是不太好的办法。
- Group Average:这种方法看起来相对有道理一些,也就是把两个集合中的点两两的距离全部放在一起求一个平均值,相对也能得到合适一点的结果。
总的来说,一般都不太用 Single Linkage 或者 Complete Linkage 这两种过于极端的方法。整个 agglomerative hierarchical clustering 的算法就是这个样子,描述起来还是相当简单的,不过计算起来复杂度还是比较高的,要找出距离最近的两个点,需要一个双重循环,而且 Group Average 计算距离的时候也是一个双重循环。
另外,需要提一下的是本文一开始的那个树状结构图,它有一个专门的称呼,叫做 Dendrogram ,其实就是一种二叉树,画的时候让子树的高度和它两个后代合并时相互之间的距离大小成比例,就可以得到一个相对直观的结构概览。不妨再用最开始生成的那个三个 Gaussian Distribution 的数据集来举一个例子,我采用 Group Average 的方式来计算距离,agglomerative clustering 的代码很简单,没有做什么优化,就是直接的双重循环:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 | def do_clustering(nodes): # make a copy, do not touch the original list nodes = nodes[:] while len(nodes) > 1: print "Clustering [%d]..." % len(nodes) min_distance = float('inf') min_pair = (-1, -1) for i in range(len(nodes)): for j in range(i+1, len(nodes)): distance = nodes[i].distance(nodes[j]) if distance < min_distance: min_distance = distance min_pair = (i, j) i, j = min_pair node1 = nodes[i] node2 = nodes[j] del nodes[j] # note should del j first (j > i) del nodes[i] nodes.append(node1.merge(node2, min_distance)) return nodes[0] |
数据点又一千多个,画出来的 dendrogram 非常大,为了让结果看起来更直观一点,我把每个叶节点用它本身的 label 来染色,并且向上合并的时候按照权重混合一下颜色,最后把图缩放一下得到这样的一个结果(点击查看原图):
或者可以把所有叶子节点全部拉伸一下看,在右边对齐,似乎起来更加直观一点:
从这个图上可以很直观地看出来聚类的结果,形成一个层次,而且也在总体上把上个大类分开来了。由于这里我把图横过来画了,所以在需要具体的 flat cluster 划分的时候,直观地从图上可以看成竖着划一条线,打断之后得到一片“森林”,再把每个子树里的所有元素变成一个“扁平”的集合即可。完整的 Python 代码如下:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 | from scipy.linalg import norm from PIL import Image, ImageDraw def make_list(obj): if isinstance(obj, list): return obj return [obj] class Node(object): def __init__(self, fea, gnd, left=None, right=None, children_dist=1): self.__fea = make_list(fea) self.__gnd = make_list(gnd) self.left = left self.right = right self.children_dist = children_dist self.depth = self.__calc_depth() self.height = self.__calc_height() def to_dendrogram(self, filename): height_factor = 3 depth_factor = 20 total_height = int(self.height*height_factor) total_depth = int(self.depth*depth_factor) + depth_factor im = Image.new('RGBA', (total_depth, total_height)) draw = ImageDraw.Draw(im) self.draw_dendrogram(draw, depth_factor, total_height/2, depth_factor, height_factor, total_depth) im.save(filename) def draw_dendrogram(self,draw,x,y,depth_factor,height_factor,total_depth): if self.is_terminal(): color_self = ((255,0,0), (0,255,0), (0,0,255))[int(self.__gnd[0])] draw.line((x, y, total_depth, y), fill=color_self) return color_self else: y1 = int(y-self.right.height*height_factor/2) y2 = int(y+self.left.height*height_factor/2) xc = int(x + self.children_dist*depth_factor) color_left = self.left.draw_dendrogram(draw, xc, y1, depth_factor, height_factor, total_depth) color_right = self.right.draw_dendrogram(draw, xc, y2, depth_factor, height_factor, total_depth) left_depth = self.left.depth right_depth = self.right.depth sum_depth = left_depth + right_depth if sum_depth == 0: sum_depth = 1 left_depth = 0.5 right_depth = 0.5 color_self = tuple([int((a*left_depth+b*right_depth)/sum_depth) for a, b in zip(color_left, color_right)]) draw.line((xc, y1, xc, y2), fill=color_self) draw.line((x, y, xc, y), fill=color_self) return color_self # use Group Average to calculate distance def distance(self, other): return sum([norm(x1-x2) for x1 in self.__fea for x2 in other.__fea]) \ / (len(self.__fea)*len(other.__fea)) def is_terminal(self): return self.left is None and self.right is None def __calc_depth(self): if self.is_terminal(): return 0 return max(self.left.depth, self.right.depth) + self.children_dist def __calc_height(self): if self.is_terminal(): return 1 return self.left.height + self.right.height def merge(self, other, distance): return Node(self.__fea + other.__fea, self.__gnd + other.__gnd, self, other, distance) def do_clustering(nodes): # make a copy, do not touch the original list nodes = nodes[:] while len(nodes) > 1: print "Clustering [%d]..." % len(nodes) min_distance = float('inf') min_pair = (-1, -1) for i in range(len(nodes)): for j in range(i+1, len(nodes)): distance = nodes[i].distance(nodes[j]) if distance < min_distance: min_distance = distance min_pair = (i, j) i, j = min_pair node1 = nodes[i] node2 = nodes[j] del nodes[j] # note should del j first (j > i) del nodes[i] nodes.append(node1.merge(node2, min_distance)) return nodes[0] |
agglomerative clustering 差不多就这样了,再来看 divisive clustering ,也就是自顶向下的层次聚类,这种方法并没有 agglomerative clustering 这样受关注,大概因为把一个节点分割为两个并不如把两个节点结合为一个那么简单吧,通常在需要做 hierarchical clustering 但总体的 cluster 数目又不太多的时候可以考虑这种方法,这时可以分割到符合条件为止,而不必一直分割到每个数据点一个 cluster 。
总的来说,divisive clustering 的每一次分割需要关注两个方面:一是选哪一个 cluster 来分割;二是如何分割。关于 cluster 的选取,通常采用一些衡量松散程度的度量值来比较,例如 cluster 中距离最远的两个数据点之间的距离,或者 cluster 中所有节点相互距离的平均值等,直接选取最“松散”的一个 cluster 来进行分割。而分割的方法也有多种,比如,直接采用普通的 flat clustering 算法(例如 k-means)来进行二类聚类,不过这样的方法计算量变得很大,而且像 k-means 这样的和初值选取关系很大的算法,会导致结果不稳定。另一种比较常用的分割方法如下:
- 待分割的 cluster 记为 G ,在 G 中取出一个到其他点的平均距离最远的点 x ,构成新 cluster H;
- 在 G 中选取这样的点 x’ ,x’ 到 G 中其他点的平均距离减去 x’ 到 H 中所有点的平均距离这个差值最大,将其归入 H 中;
- 重复上一个步骤,直到差值为负。
到此为止,我的 hierarchical clustering 介绍就结束了。总的来说,在我个人看来,hierarchical clustering 算法似乎都是描述起来很简单,计算起来很困难(计算量很大)。并且,不管是 agglomerative 还是 divisive 实际上都是贪心算法了,也并不能保证能得到全局最优的。而得到的结果,虽然说可以从直观上来得到一个比较形象的大局观,但是似乎实际用处并不如众多 flat clustering 算法那么广泛。 🙂



终于有更新啊~~~
洋洋洒洒还是分享了很多好东西~
这个blog要广而告之!
请问一下,在matlab中可以把层次聚类图画的像这里的这个一样大吗,我也有很多个点,在matlab中只能画的和屏幕一样大,所以保存下来都看不到了。还有一个问题,matlab可以像上面一样根据每个点自己本身的标签(而不是聚类后的结果)给聚类图上色吗。最后一个问题,我看您的博课里用的都是Pyson,请问一下除了matlab是否这个语言在实现科学计算方面更好用,快捷,还是其他的比如说R语言。谢谢!
能否讲下关于affinity propagation方面的东西。
谢谢
@hell
不好意思,这个东西我也没有研究过耶。
另外一种hierarchy的cluster方法是grid-based, 用网格来做 速度快 效率高
不过那个是基于大型数据的 可以在任意一层 来观全局 并且可以避免不必要的计算
觉得韩佳炜的数据挖掘书不错 每个聚类的类别都很清楚 可以看看
我都不知道我在说神马。。。。。
这个系列好像还没有结束吧?期待下文啊。
[…] /?p=407 This entry was posted in 未分类. Bookmark the permalink. ← logistic regression parameter interpretation in weka LikeBe the first to like this post. […]
你好,做了hierarchical的clustering以后怎么去确定合适的类别数?
您好,在matlab中的cluster等等的聚类函数使用的时候对于数据点数量都有限制,比如dendrogram就只能显示30个节点,这种问题怎么解决,我是matlab初学者,还请指教!还有,你的第一个用average distance聚类的代码是自己编写的?和Matlab中的不一样啊? 初学者的问题比较幼稚,还请海涵,非常感谢!
你好,Matlab 的函数限制 30 个节点应该是因为那样画出来看起来太挤了,你如果非要画那样的图的话,可以自己实现,上面的代码应该就是用 Python 来画的。
没有看过 Matlab 的实现,不同的实现有些地方看上去不一样也没有关系吧,只要主要的算法中心还是一样的话。
博主在不在?关于最短距离法聚类的matlab编程想请教博主……在下研一
如果我对200个数据先按最短聚离进行聚类,(但是用matlab自带的聚类得到的结果就是一个树状图, 没有具体的几类)然后希望用一个阈值把数据自动聚成几类,即距离小于该阈值的聚集成一类,然后得到每一个聚类有那些编号的点,然后我希望进行下一步的筛选。
希望博主能够回复,感激!
你好,从树状结构任意地方截断就可以得到一个平坦的类别划分了。
您好~很感谢,我写了一个小的聚类的程序,但是发现matlab巨累出来的结果有点奇怪,不知道能不能请您帮我看一下?我请教了别的师兄,也没有结果。多有打扰
clc;
clear all;
close all;
%% 做出点图
X =[1,1;3,1;1,1.5;3,1.5;4,1.5;1,2;3,2;3.5,2;8,2;7.5,2.5;7,3;7.5,3;8,3;8.5,3.5;4,4;5,4;4.5,4.5;4,5;5,5;]
a=X(:,1)
b=X(:,2)
figure;
plot(a,b,’rx’);
for k=1:19
text(a(k),b(k),{k});%{k}用{}将数字转换为字符
end
%% 用最短距离进行聚类
Y = pdist(X);
Z = linkage(Y,’single’);
W = cluster(Z,’cutoff’,0.5)
figure;
H = dendrogram(Z,0);
%返回值W中将点15,16,17,18,19 聚成一类,但是他们的距离显然大于0.5啊,我不知道这是怎么回事,还请指点
貌似:
to_dendrogram内
self.draw_dendrogram(draw, depth_factor, total_height/2,
depth_factor, height_factor, total_depth)
第二个参数应该是0,从x=0开始画根,否则最后到叶节点的edge就只剩一个点而已。
Group Average 为什么要算两两距离呢,其实计算每个cluster的mean,为了避免outlier,可以计算robust mean 比如l1 mean 之类的,然后计算两个mean之间的距离,这样计算量应该减少很多,没有试验过,但是觉得效果应该也不会太差吧~
京都大学今年的研究生入学考试题…
楼主有一个example的数据吗?或者简单讲一下你用的node的数据格式?
铜球!node的数据格式
最近看到的一条路是定义一个衡量聚类后质量的方程,类似于error function,以此来优化。
比如有人定义了gap指数,以聚类后的gap指数来优化k的。
来源 Estimating the number of clusters in a data set via the gap statistic
总的来说,这条思路还是一种主观定义,和Hierarchical Clustering相比,只是实现工具不同。最最核心的问题都没有直接回答。就是最终的优化条件的定义依然是主观的或者使用到先验信息。
更抽象的自动化方法,思考起来还真是烧脑。。。不知道博主最近在这方面有什么新的心得或者见闻么?
可以看一下这个:http://freemind.pluskid.org/machine-learning/deciding-the-number-of-clusterings/
我现在是初弄这个算法,一脸茫然,还请博主不吝赐教,不甚感激!
就是Pycluster包里面的treecluster的就是hierarchical cluseting算法的一种。调用的时候需要输入的数据格式是距离矩阵,比如distances = numpy.array(
[1009,
625, 1532,
559, 1034, 650,
746, 425, 1219, 615,
389, 1407, 567, 963, 1142,
157, 963, 792, 721, 726, 482,
409, 1307, 860, 994, 1071, 296, 345,
788, 1798, 760, 1250, 1534, 395, 873, 689,
716, 456, 1275, 671, 57, 1115, 671, 1015, 1505,
632, 738, 901, 297, 319, 1059, 736, 1080, 1450, 376,
508, 1214, 424, 226, 795, 831, 670, 933, 1024, 852, 478,
]
tc=Pyluster.treecluster(distancematrix=distances,method=”a” ) #groupe average
names = [ “Bloemfontein”, “Cape Town”, “Durban”, “East London”,
“George”, “Johannesburg”, “Kimberley”, “Mmabatho”, “Graskop”,
“Oudtshoorn”, “Port Elizabeth”, “Umtata”]
那我想请教一下,这个算法返回的数结构如何生成图呢?又或是如何用上述的距离矩阵按照您上面说的方法和代码做图呢(最好是叶子节点能和节点名字想对应)?期待您的回复,谢谢!
求问楼主为啥不出Evaluations的部分啦
do_clustering()中,一开始应该算距离矩阵吧?不然的话,按现有代码,是第一对(0, 1)节点马上会合并了,因为小于’inf’。此后,(0,1)两节点间的距离定义为最小距离,但它并不是节点间的最小距离,随后分类结果会全部错误。